졸업작품을 진행하며 남긴 기록들을 블로그로 옮긴 글입니다. 따라서 블로그에는 졸업작품을 완성하기 위해 적용한 글들만 옮기려고 합니다.
전체적인 내용을 확인하시려면 아래 링크로 이동해주세요 !
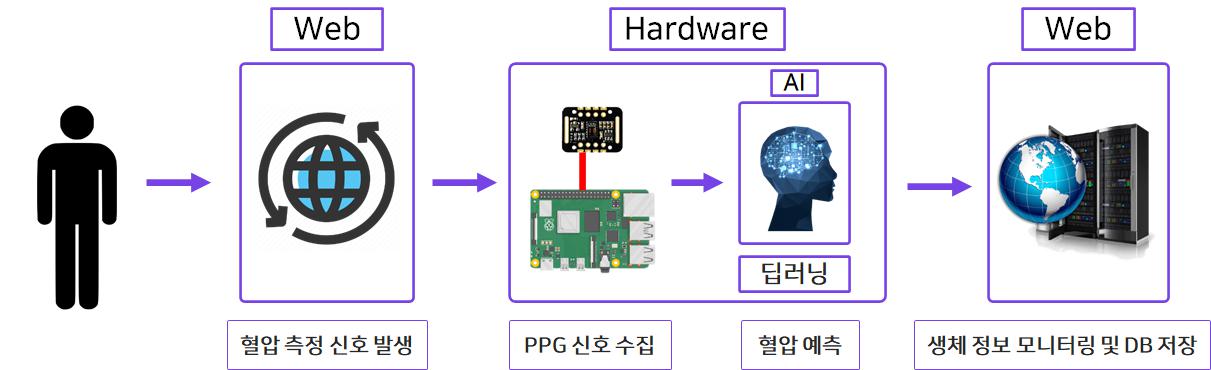
1. 개요
프로젝트 초기 구상
hardware에서 측정한 Data를Web에 전송한다.Web에서 Data를 DB에 저장하고AI를 동작시켜서 예측한 혈압 정보를 출력한다.
초기에는 논리적인 흐름에 따라 위와같이 프로젝트를 구상했다. 프로젝트를 진행할수록 Web에서 AI를 동작시키는 것이 불필요하다고 느꼈다. 차라리 hardware에서 AI를 동작시키고 SpO2, PPG, 예측 혈압 정보를 한꺼번에 Web에 전달한다면, 불필요한 동작들이 많이 제외되고 수월하게 제작할 수 있지 않을까? 라는 생각이 들었다. 마침 사용하는 라즈베리파이에서 파이썬 3.7과 텐서플로우 2.0.0이 동작하는 것을 확인했기에, 아이디어를 실현시키기로 했다.
2. 문제 발생
기존의 방식
Web → Hardware → Web → AI → Web
3. 해결 방법
새로 구상한 방식
Web → Hardware → AI → Web

A. 코드 수정
- 기존 컴퓨터로 동작하던 코드를 라즈베리파이에서 동작할 수 있게 수정해줘야한다.
- 입력되는 PPG 파일의 구성이 컴퓨터로 전처리할때와 다른 부분을 처리해야한다.
- 컴퓨터는 SpO2, BP_S, BP_D, PPG, Time 등이 나온다면, 라즈베리파이는 PPG, SpO2만 수집하기 때문이다.
- 불필요한 그래프 그리는 부분을 제거한다.
- 입력되는 PPG 파일의 구성이 컴퓨터로 전처리할때와 다른 부분을 처리해야한다.
- Web에서 얻은 데이터(키, 몸무게)를 사용하는 연결부를 함수로 만들어야 한다.
- Web으로부터 키와 몸무게를 받아서 csv 파일로 저장하게 만들었다.
- 그래서 이 부분은 별도의 함수로 만들어서 csv 파일을 간편하게 읽을 수 있게 처리하였다.
- prediction Data(수축기 혈압, 이완기 혈압)을 반환해야 한다.
- 기존에는 return을 주지 않고, 콘솔에 print하는 방식으로 진행했다.
- 하지만 예측한 혈압 데이터를 Web에 전송해야하기 때문에 예측한 혈압 데이터를 반환하는 방식으로 변경하였다.
- learn 부분은 제외한다.
- 라즈베리파이에서는 따로 학습을 하지 않고, 컴퓨터에서 학습한 모델을 불러와서 사용할 것이다.
- 그래서 learn() 함수는 사용하지 않는다.
a. 기존 컴퓨터로 동작하던 코드를 라즈베리파이에서 동작할 수 있게 수정해줘야한다.
rp_preprocess()
def rp_preprocess(self, file_name, info_dir): """ 라즈베리파이에서 수집한 데이터 전처리 :return: """ read_path = self.set_path(file_name) info_path = self.set_path(info_dir) spo2_wave_first = [5, 70] # spo2_wave_start = [] select_wave_pd = pd.DataFrame() spo2_wave_all_avg_list = [] select_wave_full_avg_list = [] ppg_pd = self.load_collection_data(path=read_path) info_pd = self.load_collection_data(path=info_path) # ppg_pd = ppg_pd[['PPG']] # ppg_tp = ppg_pd.transpose() # ppg_pd = ppg_tp.transpose() # print(ppg_pd) first_interval = ppg_pd.iloc[spo2_wave_first[0]: spo2_wave_first[1], :] # print(first_interval) first_ppg = first_interval.iloc[:,0] first_index = first_ppg.astype(float).idxmax() # spo2_wave_start.append(first_index) ppg_pd = ppg_pd.iloc[first_index:, :] spo2_wave_all = 0 for spo2_wave in ppg_pd.iloc[:, 0]: spo2_wave_all = spo2_wave + spo2_wave_all spo2_wave_all_avg = spo2_wave_all / len(ppg_pd.iloc[:, 0]) # x = range(len(ppg_pd.iloc[:, 0])) # y = [spo2_wave_all_avg for _ in x] min = ppg_pd.iloc[:, 0].max() min_1_count = 0 j_over = 0 for j in range(len(ppg_pd.iloc[:, 0])): if min > ppg_pd.iloc[j, 0]: min = ppg_pd.iloc[j, 0] if min_1_count > 0: j_over = min_1_count min_1_count = 0 else: min_1_count = min_1_count + 1 if min_1_count == 20: min_1 = ppg_pd.iloc[:j, 0].min() min_1_idx = ppg_pd.iloc[:j, 0].astype(float).idxmin() min = ppg_pd.iloc[:, 0].max() min_1_count = 0 j = j - 20 + j_over break max = ppg_pd.iloc[:, 0].min() max_1_count = 0 k_over = 0 for k in range(len(ppg_pd.iloc[:, 0])): if max < ppg_pd.iloc[j + k, 0]: max = ppg_pd.iloc[j + k, 0] if max_1_count > 0: k_over = max_1_count max_1_count = 0 else: max_1_count = max_1_count + 1 if max_1_count == 10: max_1 = ppg_pd.iloc[j: j+k, 0].max() max_1_idx = ppg_pd.iloc[j: j+k, 0].astype(float).idxmax() max = ppg_pd.iloc[:, 0].min() max_1_count = 0 k = k - 10 + k_over break for l in range(len(ppg_pd.iloc[:, 0])): if min > ppg_pd.iloc[j + k + l, 0]: min = ppg_pd.iloc[j + k + l, 0] if min_1_count > 0: min_1_count = 0 else: min_1_count = min_1_count + 1 if min_1_count == 20: min_2 = ppg_pd.iloc[j + k: j + k + l, 0].min() min_2_idx = ppg_pd.iloc[j + k: j + k + l, 0].astype(float).idxmin() min_1_count = 0 break max = ppg_pd.iloc[:, 0].max() min = ppg_pd.iloc[:, 0].min() one_wave_len = min_2_idx - min_1_idx start_point = [ppg_pd.index[0]] end_point = [start_point[0] + 127] # print(ppg_pd) x = range(len(ppg_pd.loc[:,'PPG'])) """ 환자 감시 장치에서 2분동안 수집한 PPG 신호 전체 """ # plt.rcParams["figure.figsize"] = (18, 7) # plt.plot(x, ppg_pd.loc[:,'PPG']) # # plt.axis([0, len(ppg_pd.loc['SpO2 Wave']), ppg_pd.loc['SpO2 Wave'].min(), # # ppg_pd.loc['SpO2 Wave'].max()]) # # plt.savefig(plt_save_path + "\\{0}_1_Full.png".format(num_1_file_name)) # # plt.show() # # plt.cla() """ -250 ~ 250 박스 """ y = -250 + spo2_wave_all_avg y = [y for _ in x] plt.plot(x, y, c='k') y = 250 + spo2_wave_all_avg y = [y for _ in x] plt.plot(x, y, c='k') # plt.savefig(plt_save_path + '\\{0}_6_wave_all_avg_line.png'.format(num_1_file_name)) # plt.show() # plt.cla() """ 파형 한개의 크기를 표시 빨간색 점 = 시작하는 지점 파란색 점 = 파형의 최고점 초록색 점 = 파형의 끝점 """ # min 1 plt.scatter(min_1_idx, min_1, c = 'r') # max 1 plt.scatter(max_1_idx, max_1, c = 'b') # min 2 plt.scatter(min_2_idx, min_2, c = 'g') color = ["b", "g", "r", "c", "m", "y"] for m in end_point: if m + one_wave_len < ppg_pd.index[-1]: check_point = m check_point_end = check_point + one_wave_len min_wave = ppg_pd.loc[check_point:check_point_end, 'PPG'].min() min_wave_idx = ppg_pd.loc[check_point:check_point_end, 'PPG'].astype(float).idxmin() check_point = min_wave_idx check_point_end = check_point + (one_wave_len // 1) max_wave = ppg_pd.loc[check_point:check_point_end, 'PPG'].max() max_wave_idx = ppg_pd.loc[check_point:check_point_end, 'PPG'].astype(float).idxmax() start_point.append(max_wave_idx) if max_wave_idx + 127 < ppg_pd.index[-1]: end_point.append(max_wave_idx + 127) else: end_point.append(ppg_pd.index[-1]) # plt.plot(x, ppg_pd.loc[:, "PPG"], c='k') for n in range(len(start_point[:-1])): # print(len(start_point)) x = range(start_point[n], end_point[n] + 1) y = ppg_pd.loc[start_point[n]:end_point[n], 'PPG'] # print(len(x)) # print(len(y)) plt.plot(x, y, c=color[n%len(color)]) bio_index = [] for p in range(128): bio_index.append(p) select_wave_one_avg_list = [] for o, wave_start in enumerate(start_point[:-1]): select_wave_one = copy.deepcopy(ppg_pd.loc[wave_start:end_point[o], 'PPG']) height = info_pd.iloc[0, 0] weight = info_pd.iloc[0, 1] h_w_Seris = pd.Series({'height': height, 'weight': weight}) select_wave_one_one = 0 for r in select_wave_one: select_wave_one_one = select_wave_one_one + r select_wave_one_avg = select_wave_one_one / len(select_wave_one) if max in select_wave_one.unique(): continue if min in select_wave_one.unique(): continue # if ((select_wave_one_avg - spo2_wave_all_avg) > 250) or ((select_wave_one_avg - spo2_wave_all_avg) < -250): # continue select_wave_one_avg_list.append(select_wave_one_avg) x = range(wave_start - start_point[0], wave_start + 128 - start_point[0]) y = [select_wave_one_avg for _ in x] # # plt.plot(x, select_wave_one, c='cornflowerblue') # cornflowerblue #steelblue plt.plot(x, y, c=color[o % len(color)]) for s in range(len(select_wave_one)): select_wave_one.iloc[s] = select_wave_one.iloc[s] - spo2_wave_all_avg for q in range(len(select_wave_one)): select_wave_one.iloc[q] = select_wave_one.iloc[q] / 1024 select_wave_one.index = bio_index select_wave_one = pd.concat([h_w_Seris, select_wave_one]) select_wave_pd = select_wave_pd.append(select_wave_one, ignore_index=True) plt.show() select_wave_full_avg_list.append(select_wave_one_avg_list) select_wave_pd.to_csv("rp_text.csv", mode='w', header=True)
b. Web에서 얻은 데이터(키, 몸무게)를 사용하는 연결부를 함수로 만들어야 한다.
load_collection_data()
def load_collection_data(self, path): """ 수집데이터를 불러옴 불러오는 데이터의 형식은 csv csv파일을 pandas형식으로 저장 :param path: 데이터가 저장된 위치 :return: pandas 자료형 데이터 """ read_data = pd.read_csv(path, index_col=0) return read_data
c. prediction Data(수축기 혈압, 이완기 혈압)을 반환해야 한다.
predict()
def predict(self, dir_name): """ 인공지능 모델 예측을 위한 함수 기존에 학습한 모델 정보를 통해 새로운 데이터(PPG)의 혈압 정보(수축기 혈압, 이완기 혈압)를 예측함 :param dir_name: 예측할 데이터가 있는 디렉터리명 ex) data\\unknown.csv :return: """ path = self.set_path(dir_name) csv_data = self.load_collection_data(path=path) wave = csv_data.iloc[:,0:128] HW = csv_data.iloc[:,-2:] wave_list = wave.values.tolist() Height = HW.iloc[:, 0] Weight = HW.iloc[:, 1] Height = Height.values Weight = Weight.values Height_gray_code_list = self.convert_DEC_to_GrayCode(Height) Weight_gray_code_list = self.convert_DEC_to_GrayCode(Weight) HW_gray_code_list = self.list_append(Height_gray_code_list, Weight_gray_code_list) X_np = self.list_append(wave_list, HW_gray_code_list) X_data = self.make_np_array(X_np) model = self.load_model() model.summary() print(model.predict(X_data[1:2], batch_size=self.batch_size)) predict = model.predict(X_data[:], batch_size=self.batch_size) print("X_test[1] : \n", X_data[0:10]) print("model.predict(X_test[1:2], batch_size=5) : \n", predict) BP_D_dec, BP_S_dec = self.postprocess(predict) print("\n====predict 값====\n") print("BP_D_dec", BP_D_dec) print("BP_S_dec", BP_S_dec) # for i in range(len(BP_D_dec)): print("\n====predict 값====\n") print("BP_D_dec", BP_D_dec[i]) print("BP_S_dec", BP_S_dec[i]) X_test_Series = pd.DataFrame(X_data) return BP_D_dec, BP_S_dec
4. 결론
생각나는대로 구상하고 구현하는 방식에서 조금만 생각을 해보니 더 효율적인 방법을 찾았다. 이로써 AI와 Hardware 연동 작업은 마무리되었다. 이제 남은 것은 AI 모델에 대한 마무리 작업과 문서화 부분이다.